> 2 + 2[1] 4RWie in der Beispielanwendung bereits gezeigt, besteht der Umgang mit R vor allem in der Benutzung von verschiedenen Befehlen. Die Befehle bzw. Anweisungen, werden R auf der Kommandozeile übergeben, R liest die Anweisungen ein und führt entsprechende Operationen durch. Im einfachsten Fall, kann R beispielsweise als ein überproportionierter Taschenrechner verwendet werden. In RStudio ist die Kommandozeile üblicherweise unten links zu sehen und signalisiert durch das Prompt > die Bereitschaft, Befehle annehmen zu können. Beispielsweise führt der folgende Befehl 2 + 2 auf der Kommandozeile gefolgt von einem ENTERENTER, zu folgender Ausgabe:
> 2 + 2[1] 4Das [1] kennzeichnet die erste Zeile der von R generierten Ausgabe.
Die Kommandozeile in R funktioniert nach dem Prinzip einer sogenannten REPL. REPL ist eine Abkürzung für die englischen Begriffe read-eval-print loop. Die Eingabe wird durch R eingelesen (R), im Rahmen der Programmiersprache evaluiert (E), das Ergebnis wird ausgegeben (P) und anschließend geht die Kommandozeile zurück zum Ausgangszustand (L). D.h. im Beispiel eben liest R die Eingabe 2 + 2, und evaluiert diese Anweisung. Die Evaluation führt zu dem Ergebnis 4, und das Ergebnis wird wieder auf der Kommandozeile ausgegeben. Anschließend geht die Kommandozeile zurück in den Ausgangszustand und wartet auf die nächste Eingabe >. Wenig überraschend, führt die Eingabe 3 * 3 zu folgender Ausgabe.
> 3 * 3[1] 9Da längere Analysen selten nur auf der Kommandozeile durchgeführt werden, sondern auch Skripte im Editor benutzt werden, gibt es in RStudio gibt es ein Tastenkürzel, um den Fokus auf die Kommandozeile zu setzen STRG+2STRG+2.
In der Kommandozeile in R ist es nach der Ausführung einer Anweisung nicht möglich mit dem Cursor wieder nach oben zu gehen und eine fehlerhaften Eingabe zu berichtigen. Sondern die komplette Anweisung muss noch einmal eingegeben werden. Allerdings merkt sich RStudio die Befehle und mit den Pfeiltasten UPUP und DOWNDOWN können diese wieder aufgerufen werden und entsprechend angepasst werden.
Die Anweisungen 2 + 2 oder 3 * 3 werden allgemein als Ausdrücke bezeichnet. Wir könnten die Beispiele jetzt mit den üblichen Grundrechenarten +-/* weiterführen, aber es würde nichts Neues dazukommen. Daher schauen wir uns jetzt an, wie wir komplexere, mehrstufige Berechnungen durchführen können. Dazu benötigen wir ein zentrales Konzept von Programmiersprachen: Variablen.
RNehmen wir an, wir wollen das Ergebnis der letzten komplexen Berechnung in irgendeiner Form weiter verwenden. Die im Beispiel berechnete 9 steht nach der Ausgabe allerdings für die weitere Bearbeitung nicht mehr zur Verfügung. R hat die REPL ausgeführt, die Berechnung der 9, und da mit der Ausgabe nichts Weiteres durchgeführt wurde, ist die Ausgabe auch nirgends gespeichert worden. Die Ausgabe bzw. das Ergebnis eines Befehls wird als Rückgabewert bezeichnet. Um den Rückgabewert eines Ausdrucks weiter zu bearbeiten, muss dieser Wert in irgendeiner Form in R gespeichert werden. Um Werte weiter verwenden zu können, wird den Werten daher ein Bezeichner , ein Name, zugewiesen. Es wird eine Variable eingeführt. Erfahrungsgemäß stellt dieses Konzept eine erste größere Hürde im Umgang mit R dar. Im Rahmen von Tabellenkalkulationsprogrammen spielen Variablen selten eine Rolle. Letztendlich ist eine Variable aber nichts anderes als ein Wert in R dem ein Bezeichner zugewiesen wurde.
Definition 2.1 (Variable ) Eine Variable ist ein Symbol oder ein Bezeichner, der verwendet wird, um einen Wert oder eine Datenstruktur zu speichern. Eine Variable dient als Referenz auf diesen Wert. Über den Bezeichner kann der Wert jederzeit abgerufen oder auch verändert werden.
Um in R einem Ausdruck bzw. dessen Rückgabewert einen Namen zuzuweisen, wird ein spezieller, in R definierter Befehl verwendet, der Zuweisungsoperator <-. Soll beispielsweise das Ergebnis der „komplexen“ Berechnung 3 * 3 später weiter verwendet werden, dann kann mit Hilfe des Zuweisungsoperators <- dem Rückgabewert ein Bezeichner gegeben werden.
> wert_1 <- 3 * 3R gibt jetzt keinen Ausdruck mehr zurück, da das Ergebnis des Zuweisungsoperators die Zuweisung eines Namens ist. Diese Operation gibt keinen Wert zurück. Intern hat R die Berechnung durchgeführt und dem Rückgabewert den Namen wert_1 zugewiesen. Der Name ist dabei, im Rahmen bestimmter Konventionen, vollkommen willkürlich und R hätte mich nicht daran gehindert wert_2, wert_123, thomas, steffie oder einen anderen Namen zu verwenden.
Die aktuell definierten Bezeichner sind in RStudio oben rechts unter dem Reiter Environment zu sehen.
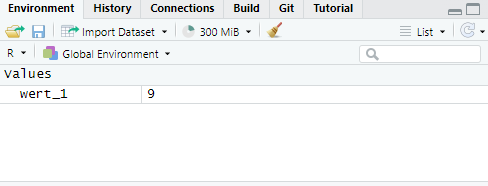
Der Wert kann nun über den Bezeichner wiederverwendet werden. D.h. die Eingabe des Namens wert_1 auf der Kommandozeile führt dazu, dass R den Wert zurückgibt. Der Wert und der Bezeichner sind nun gleichwertig miteinander. Überall wo ich einen Wert, z.B. \(9\), verwenden kann, kann ich auch den Bezeichner einsetzen.
> wert_1[1] 9Dieser Prozess funktioniert genauso mit einem komplexeren Ausdruck wie 2 + 2 * 4.
> x <- 2 + 2 * 4Der Aufruf des Bezeichners x von auf der Kommandozeile führt dann entsprechend wieder zu der Ausgabe des Wertes.
> x[1] 10Konzeptionell stellt sich der Vorgang der Zuweisung in etwa so dar. Im internen Speicher von R wird der Wert \(10\) an einer passenden Stelle abgespeichert und in einer Tabelle wird ein Eintrag mit dem Bezeichner x zusammen mit der Adresse des Wertes \(10\) abgelegt. Wenn R dann auf den Bezeichner x trifft, dann schaut es in der Tabelle nach, an welcher Stelle sich der Wert befindet und gibt diesen aus. Dies hat zur Folge, dass Bezeichner genauso wieder in Ausdrücken verwendet werden können wie Werte. R ersetzt den jeweiligen Bezeichner durch den hinterlegten Wert und führt den Ausdruck aus.
> x + wert_1[1] 19R ersetzt den Bezeichner x mit dem Wert \(10\) und den Bezeichner wert_1 mit dem Wert \(9\) und addiert die beiden Werte zusammen.
In dieser Vorgehensweise besteht ein grundlegender Unterschied zur der Arbeitsweise mit Tabellenprogrammen, bei denen immer direkt auf den jeweiligen Zellen gearbeitet wird. In R werden Berechnungen, die Rückgabewerte von Ausdrücken, Bezeichner zugewiesen, und können dann in späteren Ausdrücken (Befehlen) wieder aufgerufen werden. Andersherum, wenn Zwischenergebnisse keinen Bezeichner haben, können sie auch nicht wiederverwendet werden.
Zwei weitere Erläuterungen zu den bisherigen Beispielen sind notwendig. In den bisherigen Ausdrücken sind Leerzeichen zwischen die einzelnen Teile der Ausdrücke gesetzt worden. Diese Leerzeichen dienen lediglich der Leserlichkeit und haben keinen Einfluss auf die Evaluierung des Ausdrucks durch R. Daher sind die Ausdrücke 2 + 2 * 4 und 2+2*4 äquivalent und führen zum gleichen Ergebnis. Bei der Ausgabe des Wertes ist wahrscheinlich auch aufgefallen, dass R nicht den Wert \(16\) berechnet hat, der korrekt wäre, wenn die Evaluierung des Ausdrucks streng von links nach rechts durchgeführt würde: 2 + 2 * 4 = 4 * 4 = 16. R hat jedoch mathematische korrekt “Punkt vor Strich” angewendet und ist daher zum richtigen Ergebnis 10 gekommen.
In R wird bei Bezeichnern zwischen Groß- und Kleinschreibung unterscheidet. Daher führt der Aufruf des Bezeichners X zu einem Fehler.
> XError in eval(expr, envir, enclos): Objekt 'X' nicht gefundenDie Fehlermeldung von R gibt auch direkt an, was das Problem ist, das nämlich kein Objekt mit der Bezeichnung X gefunden werden kann.
Das Auftreten von Fehler führt bei R Neueinsteigerinnen oft zu großer Verwirrung ist aber im Programmieralltag ein vollkommen normales Ereignis und sollte daher niemanden aus der Ruhe bringen. Im vorliegenden Fall bemängelt R lediglich das es den Bezeichner X nicht finden kann und dementsprechend nicht weiß wie es weiter verfahren soll.
Es fehlt noch das letzte Konzept zu Variablen. Variablen heißen Variablen weil sie, zumindest in R variabel sein können. Es gibt keine Regel dagegen einen Bezeichner einen neuen Wert zuzuweisen. Oder anderes herum, einem anderen Wert einen zuvor verwendeten Bezeichner zu zuweisen.
> bezeichner_1 <- 1
> bezeichner_1[1] 1> bezeichner_1 <- 2
> bezeichner_1[1] 2R verhindert dies nicht und es ist die Aufgabe der Programmiererin sicherzustellen das die Bezeichner zu den intendierten Werten passen. Damit haben wir jetzt das erste große Konzept in R, bzw. in allen Programmiersprachen, das der Variable kennengelernt. Als nächstes lernen wir die verschiedenen Datentypen die Variablen in R annehmen können kennen.
Um mit R effektiv zu arbeiten ist ein zumindest oberflächliches Verständnis von sogenannten Datentypen notwendig. Konzeptionell sind Datentypen Eigenschaften von Werten die bestimmen welche Operationen mit diesem Wert möglich sind.
Definition 2.2 (Datentypen ) Datentypen sind grundlegende Kategorien, die die Art der Daten definieren, die eine Variable speichern kann. Sie bestimmen, welche Operationen auf den Daten ausgeführt werden können und wie sie in R verarbeitet werden.
Der einfachste Datentyp numerisch erlaubt zum Beispiel die üblichen mathematischen Operationen +-/* die wir bereits kennengelernt haben. Eine Zeichenkette "Haus" lässt sich dagegen nicht dividieren. Die Werte können auch als Objekte bezeichnet werden, was konzeptionell einfacher nachzuvollziehen. Den Datentypen eines Objektes kann mittels der Funktion typeof() bestimmt werden. Ein Objekt hat einen Typ und einen Wert. Wir beginnen mit den grundlegendsten, den atomaren (atomic), Datentypen: Numerisch, Zeichenketten und logische Werte.
Wie bereits beschreiben, ist einer der einfachsten Datentypen ein numerischer Wert wie 1, 123345 12.3456. Es gibt noch eine Unterscheidung in R zwischen ganzzahligen Werten (integer) und Dezimalzahlen (double), wobei für die meisten Anwendungsfälle die Unterscheidung nicht von großer Bedeutung ist. Das Dezimaltrennzeichen in R ist ein Punkt ..
> dezimal_zahl <- 12.345
> dezimal_zahl[1] 12.345> typeof(dezimal_zahl)[1] "double"> typeof(12.345)[1] "double"Integer Werte werden in R mit einem angehängtem L spezifiziert. Wir kein L angehängt geht R per default bei Zahlen immer von einer Dezimalzahl aus.
> ganz_zahl <- 12345L
> ganz_zahl[1] 12345> typeof(ganz_zahl)[1] "integer"> typeof(12345)[1] "double"> typeof(12345L)[1] "integer"Die numerischen Werte erlauben die üblichen mathematischen Operationen.
Der nächste Datentyp sind Zeichenketten (character in den meisten anderen Programmiersprachen strings). Zeichenketten repräsentieren Textdaten und werden oft für die Manipulation von Texten und die Verarbeitung von Zeichen verwendet. Zeichenketten können mit einfachen Anführungszeichen (') oder doppelten Anführungszeichen (") erstellt werden.
> s_1 <- "Haus"
> s_1[1] "Haus"> typeof(s_1)[1] "character"In R wird der Typ von Zeichenketten als character bezeichnet.
Bezüglich der Anführungszeiche, besteht semantisch kein Unterschied zwischen den einfachen und den doppelten Anführungszeichen und es ist mehr ein Zeichen persönlicher Präferenz welche Art benutzt wird. Ein Anwendungsfall wo die Art von Bedeutung ist, erfolgt wenn innerhalb der Zeichenketten Anführungszeichen benötigt werden. Dann müssen die äußeren Zeichenketten der jeweils andere Typ sein, da ansonsten R die Zeichenkette nicht als solche erkennt. Also zum Beispiel wenn ich als Zeichenkette den Wert: Er sagte: "Nein"!, benötige, verwende ich die einfachen Anführungszeichen um R zu signalisieren das ich eine Zeichenkette benötige.
> s_2 <- 'Er sagte: "Nein"!'
> s_2[1] "Er sagte: \"Nein\"!"Um Operationen auf Zeichenketten anzuwenden gibt es in R eine ganze Reihe von spezialisierten Funktionen. Möchte ich zum Beispiel einen Teil der Zeichen aus einer Zeichenkette extrahieren, kann ich die substring()-Funktion verwenden.
> s_3 <- "DasisteinelangeZeichenkette"
> substring(s_3, 4, 6)[1] "ist"Der erste Parameter übergibt die Zeichenkette an substring() während der zweiter Parameter den Startposition und der dritte Parameter die Endposition des zu extrahierenden Strings angibt.
Die Länge einer Zeichenkette kann mit der Funktion nchar() bestimmt werden.
> nchar(s_3)[1] 27Eine Funktion die oft mit Zeichenketten eine Anwendung findet ist die paste() bzw. die Spezialform paste0(). Mit paste() können Zeichenkette zusammengesetzt werden.
> paste('Ich', 'bin', 'ein', 'Berliner')[1] "Ich bin ein Berliner"Per default fügt paste() ein Leerzeichen zwischen die Zeichenketten ein. Dies kann entsprechend angepasst werden.
> paste('Ich', 'bin', 'ein', 'Kölner', sep = '--')[1] "Ich--bin--ein--Kölner"Die Argumente an paste() müssen nicht alle Zeichenketten sein, werden aber dann in Zeichenketten konvertiert.
> paste(3, 2, '-mal', sep='')[1] "32-mal"paste0() ist eine Spezielform von paste() bei der das Argument sep auf "" gesetzt ist. Das heißt, die Zeichenketten werden direkt aneinander gehängt.
> paste0('kein','zwischen','raum')[1] "keinzwischenraum"Das Paket stringr bietet eine große Sammlung von Funktion die den Umgang und die Manipulation von Zeichenketten stark vereinfachen.
Der nächste Datentyp sind sogenannte logische Werte oder Wahrheitswerte. Logische Werte können nur einen von zwei Werten annehmen, entweder WAHR oder FALSCH. Logische Ausdrücke kennt ihr wahrscheinlich aus der Schule in Form von Wahrheitstabellen bei denen boolesche Werte entweder mit und \(\cap\) oder oder \(\cup\) verknüpft werden (siehe Tabelle 2.1).
| \(\cap\) | TRUE | FALSE |
|---|---|---|
| TRUE | TRUE | FALSE |
| FALSE | FALSE | FALSE |
| \(\cup\) | TRUE | FALSE |
|---|---|---|
| TRUE | TRUE | TRUE |
| FALSE | TRUE | FALSE |
In R werden die beiden Werte mit TRUE und FALSE oder in der abgekürzten Form T und F dargestellt.
> wahr <- TRUE
> wahr[1] TRUE> falsch <- FALSE
> falsch[1] FALSEDie beiden Verknüpfungen werden mit & für \(\cap\) und | für \(\cup\).
> wahr & wahr[1] TRUE> wahr & falsch[1] FALSE> falsch & falsch[1] FALSE> wahr | wahr[1] TRUE> wahr | falsch[1] TRUE> falsch | falsch[1] FALSELogische Werte sind vor allem bei der Ablaufkontrolle und beim Indexieren wichtig.
Logische Werte können in numerische Werte konvertiert (coercion) werden, dabei wird FALSE zu \(0\) und TRUE zu \(1\). Mit der Funktion as.numeric() können wir zum Beispiel Objekte von einem Typ in einen numerischen Typ konvergieren.
> as.numeric(wahr)[1] 1> as.numeric(falsch)[1] 0Die umgekehrte Richtung, von numerisch zum logischen Wert, kann mittels der Funktion as.logical() durchgeführt werden. Dabei werden alle Werte \(\neq0\) zu WAHR und \(0\) zu FALSCH.
> as.logical(123)[1] TRUE> as.logical(1.23)[1] TRUE> as.logical(0)[1] FALSEDie Konvertierung von einem Datentyp in einen anderen Datentyp passiert zum Teil auch automatisch. Gebe ich zum Beispiel den foglenden Ausdruck ein:
> 1 & TRUE[1] TRUEErhalten wir als Ergebnis des Ausdrucks den Wert TRUE. Der Operator & kann nur mit logischen Werten arbeiten. Wir haben aber einen numerischen und einen logischen Wert übergeben. R versucht daher zunächst den nicht-logischen Wert in einen logischen Wert zu konvertieren. In dem Falle geht das problemlos da numerische Werte, wie oben gesprochen, nach der folgenden Regel konvertiert werden.
\[\begin{equation*} \text{logical}(x) = \begin{cases} \text{F}, \text{if } x = 0 \\ \text{T}, \text{if } x \neq 0 \end{cases} \end{equation*}\]
Nach dieser Regel wird die 1 in TRUE konvertiert und dann der neue Ausdruck TRUE & TRUE ausgewertet, der dann entsprechend zu TRUE führt.
Eine praktische Funktion im Zusammenhang ist logischen Werte, insbesonderen logischen Vektoren, ist which(). which() gibt bei einen logischen Vektor die Position der Wert mit dem Wert TRUE.
> which(c(TRUE, FALSE, TRUE, FALSE, TRUE, TRUE))[1] 1 3 5 6Vektoren sind ein grundlegender Datentypein R und können sowohl numerische als auch nicht-numerische Werte speichern. Allerdings kann immer nur eine Datentyp in einem Vektor gespeichert werden. Da Vektoren sehr oft bei Datenanalysen verwendet werden und oft zur Datenrepräsentation verwendet werden, werden sie entsprechend direkt in R unterstützt. Etwas allgemeiner betrachtet können Vektoren als eine geordnete Sammlung von Elementen betrachtet werden. Geordnet bedeutet dabei, dass sie ein feststehende Abfolge haben.
Die direkteste Art einen Vektoren in R zu erstellen, ist mittels der c() Funktion (c von combine). Wollen wir zum Beispiel einen numerischen Vektor mit den folgenden Einträgen erstellen.
\[\begin{equation} v_1 = \begin{pmatrix}3\\7\\8\end{pmatrix} \end{equation}\]
Dann sieht dies in R folgendermaßen aus.
> v_1 <- c(3,7,8)
> v_1[1] 3 7 8Das gleiche Prinzip funktioniert auch mit anderen Typen. Beispielweise mit einem Zeichenkettenvektor.
> v_2 <- c('mama','papa','tochter')
> v_2[1] "mama" "papa" "tochter"Oder einem logischen Vektor.
> v_bool <- c(TRUE, TRUE, FALSE, TRUE)Auf einzelne Elemente oder Teile eines Vektor kann mittels des sogenannten subsettings zugegriffen werden. Jeder Vektor hat eine definierte Länge die wir mit length() bestimmen können.
> length(v_2)[1] 3Mit eckigen Klammern [] können wir Teilelemente des Vektors extrahieren. Die Indizierung geht dabei von \(1\) bis $n = $ length(Vektor). Wollen wir zum Beispiel das zweite Element aus \(v_1\) extrahieren, dann verwenden wir.
> v_1[2][1] 7Wenn der Index außerhalb des erlaubten Bereichs ist, dann wird ein NA für (N)ot (A)vailable von R zurückgegeben.
> v_1[10][1] NAR erlaubt die Verwendung von Vektoren zum subsetten. Wenn ich das 1. und 3. Element aus \(v_2\) extrahieren möchte, kann ich einen Vektor mit den beiden Elementen \(1\) und \(3\), \(v_{\text{index}} = (1,3)\) an [] übergeben.
> v_i <- c(1,3)
> v_2[v_i][1] "mama" "tochter"Das funktioniert ebenfalls ohne den Zwischenvektor.
> v_2[c(1,3)][1] "mama" "tochter"Der zurückgegebene Wert ist dann auch wieder ein Vektor. Wir können dadurch zum Beispiel einen Vektor erstellen, der länger als der Ursprungsvektor ist.
> v_1[c(1,1,2,2,3,3)][1] 3 3 7 7 8 8Zum subsetten können auch logische Vektoren verwendet werden. Der resultierende Vektor ist ein Vektor mit denjenigen Elementen, für die der logische Vektor den Wert TRUE hat.
> v_bool <- c(TRUE, FALSE, TRUE)
> v_1[v_bool][1] 3 8Wird ein negativer Wert an [] übergeben, dann wird dieser bzw. diese Wert(e) von R ausgeschlossen.
> v_1[-2][1] 3 8Das funktioniert auch wieder mit Vektoren.
> v_lang <- c(1,2,3,4,5,6,7)
> v_lang[-c(2,3,6)][1] 1 4 5 7Bei all diesen Anwendungen ist immer darauf zu achten, dass solange keine Zuweisung mittels <- ausgeführt, der jeweilige Vektor nicht verändert wird.
Auf numerische Vektoren können die gängigen mathematischen Operatoren (+-*/) angewendet werden. Die Operationen werden elementweise ausgeführt.
\[\begin{equation*} \begin{pmatrix}v_1\\v_2\\\vdots\\v_n\end{pmatrix} \times \begin{pmatrix}a\\b\\\vdots\\c\end{pmatrix} = \begin{pmatrix}av_1\\bv_2\\\vdots\\cv_n\end{pmatrix} \cdot \end{equation*}\]
Dementsprechend können Vektoren auch addiert werden, wenn die Längen gleich sind. Die Addition erfolgt dann ebenfalls elementweise.
> v_3 <- c(1,2,3)
> v_4 <- c(4,5,6)
> v_3 + v_4[1] 5 7 9Multiplikation mit einem Skalar folgt der üblichen Skalarmultiplikation von Vektoren aus der Mathematik.
\[\begin{equation} \begin{pmatrix}3\\7\\8\end{pmatrix} \cdot 3 = \begin{pmatrix}9\\21\\24\end{pmatrix} \end{equation}\]
> v_1 * 3[1] 9 21 24Eine etwas unglückliche gewählte Operation von R ist, dass auch Vektoren mit unterschiedlichen Längen addiert (subtrahiert, multipliziert, dividiert) werden können. Allerdings nur wenn die Länge des längeren Vektors ein Vielfaches des kürzeren Vektors ist. R wiederholt dann die Abfolge der Elemente des kürzeren Vektors so oft bis die Länge des längeren Vektors erreicht wird.
> v_5 <- c(1,2)
> v_6 <- c(1,2,3,4,5,6)
> v_5 + v_6[1] 2 4 4 6 6 8Dieses Verhalten ist wie gesagt etwas unglücklich gewählt, da sich sehr schnell subtile Fehler in den Code einschleichen können. Diese sind erfahrungsgemäß nur sehr schwer wieder ausfindig zu machen. Daher sollte auf diese Feature möglichst verzichtet werden.
Das subsetting wird auch benutzt, wenn ich einem bestehenden Vektor Werte zuweisen will. Möchte ich zum Beispiel bei \(v_1\) den zweiten Wert von einer \(2\) in eine \(20\) ändern. Dann verwende ich wiederum [] mit dem Indexwert 2 weise den entsprechend wert 20 zu.
> v_1[2] <- 20
> v_1[1] 3 20 8Dies funktioniert auch für mehrere Werte
> v_1[1:2] <- c(10, 100)
> v_1[1] 10 100 8Ich kann auch nur einen einzelnen Wert zuweisen, der dann an mehrere Stellen geschrieben wird.
> v_1[2:3] <- 1000
> v_1[1] 10 1000 1000Das funktioniert auch mit logischen Indexvektoren.
> v_1[c(TRUE,FALSE,TRUE)] <- -13
> v_1[1] -13 1000 -13Per default werden Vektoren in R nicht als Spaltenvektoren wie in der Mathematik angesehen. Das kann manchmal ebenfalls zu Problemen in Rechnungen führen, wenn Formeln eins-zu-eins in R übertragen werden und die Formel von der üblichen Algebra bei Vektoren und Matrizen ausgeht. Dies sollte daher im Hinterkopf behalten werden.
Wenn das Skalarprodukt \(v_x v_y = \sum_{i=1}^nv_{xi}v_{yi}\) zweier Vektoren berechnet werden soll, dann muss ein spezieller Matrizenmultiplikator benutzt werden %*%. In unserem Beispiel mit \(v_3\) und \(v_4\)
\[\begin{equation*} v_3 \cdot v_4 = 1\cdot4 + 2\cdot5 + 3\cdot6 = 32 \end{equation*}\]
> v_3 %*% v_4 [,1]
[1,] 32In R gibt es verschiedene Funktion um schnell Vektoren mit bestimmten Strukturen zu erstellen. Wird ein Vektor mit aufeinanderfolgenden Ganzzahlen benötigt kann der : Operator verwendet werden. Es wird ein Vektor nach dem Muster a:b = \((a,a+1,\ldots,b-1,b)\) erstellt.
> 1:3[1] 1 2 3> 10:15[1] 10 11 12 13 14 15Soll die Zahlenfolgen nicht ganzzahlig sein sondern ein anderes Interval haben, dann kann die seq() Funktion verwendet werden.
[1] 1 3 5 7 9
[1] 0.00 0.25 0.50 0.75 1.00 1.25 1.50 1.75 2.00
[1] 0.0000000 0.6981317 1.3962634 2.0943951 2.7925268 3.4906585 4.1887902
[8] 4.8869219 5.5850536 6.2831853Eine weitere Funktion die oft einen Einsatz findet ist die rep() Funktion mit der Vektoren mit bestimmten Wiederholungsmustern erstellt werden können.
[1] 3 3 3 3 3
[1] 1 2 1 2 1 2
[1] 1 1 1 2 2 2Wie in den Beispielen gezeigt, können relativ unkompliziert Vektoren mit beliebigen Wiederholungen erzeugt werden.
Eine weitere praktische Funktion in diesem Zusammenhang ist die Funktion paste() die wir schon bei den Zeichenketten kennengelernt haben. Werden Vektoren an paste() übergeben, werden die kürzeren Werte so oft wiederholt bis die Länge des längsten Vektors erreicht wird. Dann erfolgt erst die Zusammensetzung der Vektoren wiederum elementweise. Als Rückgabewert wird daher wiederum ein Vektor gegeben.
> paste('Participant', 1:3, sep = '_')[1] "Participant_1" "Participant_2" "Participant_3"> paste(c('A','B'), 1:4, sep = 'x')[1] "Ax1" "Bx2" "Ax3" "Bx4"Zuletzt, benötige ich einen Vektor einer bestimmen Länge bei dem alle Elemente gleich sind kann ich die Funktionen numeric(), character() und logical() verwenden. Mit der Funktion numeric(n) kann ein numerischer Vektor mit \(n\) Nullen erstellt werden.
> numeric(5)[1] 0 0 0 0 0Wird zum Beispiel ein numerischer Vektor der Länge \(n=11\) mit ausschließlich \(13\) als Einträgen benötigt, dann könnte dies wie folgt erreicht werden:
> numeric(11) + 13 [1] 13 13 13 13 13 13 13 13 13 13 13Da Vektoren ein zentraler Datentyp in R sind, ist es wichtig sich möglichst eingehend mit den Eigenschaften von Vektoren zu beschäftigen. Es ist praktisch unmöglich in R produktiv zu werden ohne zumindest die Grundeigenschaften von Vektoren verstanden zu haben.
Matrizen sind rechteckige und damit zweidimensionale Datenstrukturen, die aus Zeilen und Spalten bestehen und als Verallgemeinerung von Vektoren zu verstehen sind. Matrizen liegen zahllosen Berechnungen in der Statistik zugrunde, auch wenn diese vorwiegend im Hintergrund stattfinden. In R können Matrizen genauso wie Vektoren auch Werte wie Zeichenketten oder logischen Werten haben. Wir werden uns allerdings auf numerische Matrizen beschränken, da diese am ehesten benötigt werden.
Eine Matrize der Dimension \(m \times n\) wird in der Mathematik wie folgt definiert.
\[\begin{equation} A = (a_{ij}) = \begin{pmatrix}a_{11} & \ldots & a_{1n} \\ \vdots & & \vdots \\ a_{m1} & \ldots & a_{mn} \end{pmatrix} \end{equation}\]
In R werden Matrizen mit der matrix()-Funktion erzeugt. In den meisten Fällen sind zwei von drei Parametern notwenig. Der erste Parameter gibt die einzelnen Werte an gefolgt von der Anzahl der Zeilen (nrow) und der Anzahl der Spalten (ncol). Meist sind nur zwei Parameter notwendig, da wenn z.B. \(m\times n\) Werte an matrix() übergeben werden, dann kann nach Angabe der Anzahl der Zeilen, die Anzahl der Spalten hergeleitet werden bzw. genauso andersherum.
> matrix(1:6, nrow = 3, ncol = 2) [,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6> matrix(1:6, nrow = 3) [,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6> matrix(1:6, ncol = 2) [,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6Wie immer, wenn die Matrize weiter benötigt wird, dann muss der Rückgabewert von matrix() einer Variable zugewiesen werden.
> mat_1 <- matrix(1:4, nrow=2, ncol = 2)
> mat_1 [,1] [,2]
[1,] 1 3
[2,] 2 4Um Elemente in der Matrize zu manipulieren oder zu extrahieren verwendet wir wieder den subsetting Operator []. Im Unterschied zum Vektor, der nur eine Dimension hat, müssen bei einer Matrize dzwei Indezis angegeben werden. Die Spezifikation erfolgt nach dem Muster mat[zeile,spalte]. Wie beim Vektor sind bei der Indizierung Vektoren, logische Werte und negative Werte erlaubt. Wenn ihr eine Dimension weglasst, werden entsprechend alle Zeilen bzw. Spalten angesteuert.
> mat_1[1,1] # Element a_11[1] 1> mat_1[2,2] # Element a_22[1] 4> mat_1[,1] # Elemente a_i1[1] 1 2> mat_1[2,] # Elemente a_2j[1] 2 4> mat_1[-2,1] # alle Elemente der ersten Spalte ohne Zeile 2[1] 1Über das subsetting können den indizierten Werte in der Matrize neue Werte zugewiesen werden.
> mat_0 <- matrix(0, nr=6, nc = 3)
> mat_0 [,1] [,2] [,3]
[1,] 0 0 0
[2,] 0 0 0
[3,] 0 0 0
[4,] 0 0 0
[5,] 0 0 0
[6,] 0 0 0> mat_0[2:3,2] <- 1:2
> mat_0[5,2:3] <- -13
> mat_0 [,1] [,2] [,3]
[1,] 0 0 0
[2,] 0 1 0
[3,] 0 2 0
[4,] 0 0 0
[5,] 0 -13 -13
[6,] 0 0 0Matrizen können wie Vektoren auf zwei Arten multipliziert werden. Entweder die Elementweise mittels * oder Matrizenmultiplikation über %*% nach dem Muster.
> A <- matrix(1:6, nr=2)
> B <- matrix(1:6, nr=3)
> A [,1] [,2] [,3]
[1,] 1 3 5
[2,] 2 4 6> B [,1] [,2]
[1,] 1 4
[2,] 2 5
[3,] 3 6> A %*% B [,1] [,2]
[1,] 22 49
[2,] 28 64Um die Dimension einer Matrize, d.h. die Anzahl der Zeilen und Spalten, zu bestimmen gibt es in R die Funktion dim().
> dim(A)[1] 2 3Der erste Wert gibt entsprechend die Anzahl der Zeilen an während der zweite Wert die Anzahl der Spalten anzeigt. Wenn nur einer der Werte benötigt wird, gibt es auch die beiden Kurzfunktionen:
> nrow(A)[1] 2> ncol(A)[1] 3Ansonsten funktionieren die mathematischen Operator wie in der Matrizenalgebra zu erwarten ist. Wenn z.B. zwei Matrizen addiert werden sollen, dann müssen die Dimensionen übereinstimmen.
> A + BError in A + B: nicht passende Arrays> D <- matrix(11:16, nr=2)
> dim(D)[1] 2 3> A + D [,1] [,2] [,3]
[1,] 12 16 20
[2,] 14 18 22Mit der Funktion diag() kann eine quadratische Diagonalmatrize erzeugt werden. Eine Diagonalmatrize hat nur auf der sogenannten Hauptdiagonalen Einträge, während alle anderen Werte \(=0\) sind (siehe Formel \(\eqref{eq-r-kickoff-diag}\)).
\[\begin{equation} \begin{pmatrix} d_{11}& 0 & 0 & \cdots & 0 \\ 0 & d_{22} & 0 & &\vdots \\ 0 & 0 & \ddots & & \\ \vdots& & & & \\ 0 & \cdots & & & d_{nn} \end{pmatrix} \label{eq-r-kickoff-diag} \end{equation}\]
Eine in der Statistik und generell in der linearen Algebra wichtige Matrize ist die Einheitsmatrize \(\mathbf{I}_n\). Die Einheitsmatrize ist eine Diagonalmatrize bei der alle Einträge auf der Hauptdiagonalen \(=1\) sind.
\[\begin{equation} \mathbf{I}_n = \begin{pmatrix} 1 & 0 & \cdots & 0 \\ 0 & 1 & & \vdots \\ \vdots & & \ddots & \\ 0 & \cdots & & 1 \end{pmatrix} \label{eq-unity-matrix} \end{equation}\]
Der Parameter \(n\) gibt die Dimension an. Da es sich um eine quadratische Matrize handelt ist die Anzahl der Zeilen gleich der Anzahl der Spalten. Die Einheitsmatrize hat in der linearen Algebra die Funktion des Einselement. Wenn eine Matrize \(M\) mit der Einheitsmatrize \(\mathbf{I}\) multipliziert, dann ist das Ergebnis wieder die Matrize \(M\), wie auch \(m \cdot 1 = m\).
Mit diag(n) kann eine Einheitsmatrize in R generiert werden.
> I_3 <- diag(3)
> I_3 [,1] [,2] [,3]
[1,] 1 0 0
[2,] 0 1 0
[3,] 0 0 1> M <- matrix(1:9, nr=3)
> I_3 %*% M [,1] [,2] [,3]
[1,] 1 4 7
[2,] 2 5 8
[3,] 3 6 9> M %*% I_3 [,1] [,2] [,3]
[1,] 1 4 7
[2,] 2 5 8
[3,] 3 6 9Möchte ich eine Diagonalmatrize erstellen die bestimmte Wert auf der Hauptdiagonalen hat, dann kann ich diag() auch einen Vektor mit den Werten übergeben.
> diag(1:4) [,1] [,2] [,3] [,4]
[1,] 1 0 0 0
[2,] 0 2 0 0
[3,] 0 0 3 0
[4,] 0 0 0 4Wenn diag() eine Matrize übergeben wird, dann werden die Elemente auf der Hauptdiagonalen extrahiert.
> E <- diag(1:4)
> diag(E)[1] 1 2 3 4Eine Matrize kann mit Hilfe der t() Funktion transponiert werden.
\[\begin{align*} A^T = a_{ji} = \begin{pmatrix}a_{11} & \ldots & a_{m1} \\ \vdots & & \vdots \\ a_{n1} & \ldots & a_{mn} \end{pmatrix} \end{align*}\]
Ein anschauliches Beispiel:
\[\begin{align*} (a_{ij}) &= \begin{pmatrix} 1 & 3 & 5 \\ 2 & 4 & 6\end{pmatrix} \\ (a_{ij})^T = (a_{ji}) &= \begin{pmatrix} 1 & 2 \\ 3 & 4 \\ 5 & 6 \end{pmatrix} \end{align*}\]
> t(A) [,1] [,2]
[1,] 1 2
[2,] 3 4
[3,] 5 6D.h. beim transponieren, werden die Zeilen und Spalten der Matrize vertauscht.
Mit den Funktionen cbind() und rbind() können zusätzliche Spalten bzw. Zeilen an eine Matrize angehängt werden. Dabei werden die Argumente so wiederholt, dass die Anzahl der Elemente übereinstimmt, bzw. ein Fehler geworfen wenn die Anzahl kein Vielfaches voneinander ist.
> cbind(1:2, E) [,1] [,2] [,3] [,4] [,5]
[1,] 1 1 0 0 0
[2,] 2 0 2 0 0
[3,] 1 0 0 3 0
[4,] 2 0 0 0 4> rbind(E,1:2) [,1] [,2] [,3] [,4]
[1,] 1 0 0 0
[2,] 0 2 0 0
[3,] 0 0 3 0
[4,] 0 0 0 4
[5,] 1 2 1 2Um die Inverse eine quadratischen Matrize zu berechnen bzw. um lineare Gleichungsysteme zu lösen kann die solve() Funktion verwendet werden.
> E_inv <- solve(E)
> E_inv %*% E [,1] [,2] [,3] [,4]
[1,] 1 0 0 0
[2,] 0 1 0 0
[3,] 0 0 1 0
[4,] 0 0 0 1Um das Gleichungssystem
\[\begin{align*} 2x + 3y &= 7 \\ -4x + y &= 2 \\ \end{align*}\]
zu lösen, kann zum Beispiel der folgende Code verwendet.
> A <- matrix(c(2,-4,3,1), nr=2)
> y <- c(7,2)
> x <- solve(A,y)
> x[1] 0.07142857 2.28571429> A %*% x [,1]
[1,] 7
[2,] 2Weitere Funktionen wie beispielweise die QR-Zerlegung findet ihr in der Dokumentation.
Eine weitere hilfreiche Funktion im Zusammenhang mit der Programmierung mit Matrizen ist die apply() Funktion. Mit apply() kann eine Funktion auf die einzelnen Spalten oder Zeilen einer Matrize angewendet werden.
[,1] [,2] [,3]
[1,] 1 1 1
[2,] 1 1 1
[3,] 1 1 1
[4,] 1 1 1
[1] 3 3 3 3
[1] 4 4 4data.frame() oder tibble()Dataframes sind eine zentrale Datenstruktur in R da praktisch alle Arten von Daten mittels Dataframes organisiert werden. Konzeptionell sind Dataframes ähnlich zu Matrizen, sie ermöglichen allerdings die Speicherung verschiedener Datentypen (z. B. numerisch, Zeichenfolgen, Faktoren) in verschiedenen Spalten ähnlich wie die Daten in einem Tabellenprogramm organisiert sind. Jede Spalte hat dabei die gleiche Anzahl an Elementen und muss einen eineindeutigen Namen haben.
Klassischerweise werden Dataframes mittels der Funktion data.frame() erstellt. Eine modernere Version sind die sogenannten tibbles() aus dem Package tibble. Dieses werden hier im Weiteren verwenden, d.h. tibbles() aus Anwendersicht verschiedene Verbesserungen mitbringen. Wir verwenden jedoch weiter den Term Dataframe um den Datentyp zu becshreiben. Daher, wenn wir ein Dataframe mittels tibble() erstellen wollen, müssen wir zunächst das Package tibble() laden.
> library(tibble)Dann können wir einen Dataframe erstellen.
> df_1 <- tibble(a = 1:3, b = c('eins','zwei','drei'))
> df_1# A tibble: 3 × 2
a b
<int> <chr>
1 1 eins
2 2 zwei
3 3 drei Wenn ich mir den Inhalt eines Dataframes ausgeben lasse, dann werden mir die Namen der Spalten angezeigt, in vorliegenden Fall a und b. Gefolgt vom Datentyp hier <int> den wir strenggenommen noch nicht kennengelernt haben aber auch ein numerischer Typ ist, und <chr> was die Kurzform für character und entsprechend Zeichenketten bezeichnet. Danach folgen die Einträge in den beiden Spalten. Standardmäßig werden nur die ersten zehn Zeilen gezeigt, was in diesem Fall nicht weiter auffällt.
Wir können auf die einzelnen Spalten über zwei verschiedene Operatoren zugreifen. Einmal mittels des $ Operator oder über [[]] Im ersten Fall können wir den Namen direkt verwenden.
> df_1$a[1] 1 2 3Bei der doppelten, eckigen Klammer [[]] müssen wir den Namen als eine Zeichenkette, also entweder in ' oder " einschließen.
> df_1[['a']][1] 1 2 3Wenn ich eine neue Spalte hinzufügen will, kann ich einfache das Dataframe nehmen und über die Syntax von eben eine neue Spalte definieren.
> df_1$neu <- 11:13
> df_1# A tibble: 3 × 3
a b neu
<int> <chr> <int>
1 1 eins 11
2 2 zwei 12
3 3 drei 13Entsprechend kann ich über eine Kombination von Spaltennamen und subsetting einzelne Elemente verändern.
> df_1$b[2] <- "AA"
> df_1# A tibble: 3 × 3
a b neu
<int> <chr> <int>
1 1 eins 11
2 2 AA 12
3 3 drei 13Da Daten praktisch immer in Form von Dataframes bearbeitet werden, gibt es eine ganze Reihe von Hilfsfunktionen um mit Dataframes zu arbeiten. Erstellen wir uns zunächst ein etwas größeres Beispiel.
> df_2 <- tibble(
+ id = paste0('P', 1:10),
+ gruppe = rep(c('CON','TRT'), each=5),
+ wert = 21:30
+ )
> df_2# A tibble: 10 × 3
id gruppe wert
<chr> <chr> <int>
1 P1 CON 21
2 P2 CON 22
3 P3 CON 23
4 P4 CON 24
5 P5 CON 25
6 P6 TRT 26
7 P7 TRT 27
8 P8 TRT 28
9 P9 TRT 29
10 P10 TRT 30Mit der Funktion summary() erstellt R eine kurze Zusammenfassung mit deskriptiven Statistiken zum Dataframe.
> summary(df_2) id gruppe wert
Length:10 Length:10 Min. :21.00
Class :character Class :character 1st Qu.:23.25
Mode :character Mode :character Median :25.50
Mean :25.50
3rd Qu.:27.75
Max. :30.00 Eine ähnliche Funktion erfüllt die Funktion glimpse() aus dem Package tibble().
> glimpse(df_2)Rows: 10
Columns: 3
$ id <chr> "P1", "P2", "P3", "P4", "P5", "P6", "P7", "P8", "P9", "P10"
$ gruppe <chr> "CON", "CON", "CON", "CON", "CON", "TRT", "TRT", "TRT", "TRT", …
$ wert <int> 21, 22, 23, 24, 25, 26, 27, 28, 29, 30Die ersten n Elemente (default n = 6) mit head().
> head(df_2, n = 4)# A tibble: 4 × 3
id gruppe wert
<chr> <chr> <int>
1 P1 CON 21
2 P2 CON 22
3 P3 CON 23
4 P4 CON 24Die letzten n elemente (default n = 6) mit tail().
> tail(df_2, n = 3)# A tibble: 3 × 3
id gruppe wert
<chr> <chr> <int>
1 P8 TRT 28
2 P9 TRT 29
3 P10 TRT 30Die Spaltennamen werden mittels names() ausgegeben.
> names(df_2)[1] "id" "gruppe" "wert" Die names() Funktion kann auch verwendet werden um die Spaltennamen zu verändern. Dazu kann entweder subsetting angewendet werden um bestimmte Namen zu verändern oder auch alle auf einmal.
> names(df_2)[3] <- 'value'
> df_2# A tibble: 10 × 3
id gruppe value
<chr> <chr> <int>
1 P1 CON 21
2 P2 CON 22
3 P3 CON 23
4 P4 CON 24
5 P5 CON 25
6 P6 TRT 26
7 P7 TRT 27
8 P8 TRT 28
9 P9 TRT 29
10 P10 TRT 30> names(df_2) <- c('pid', 'group', 'measurement')
> df_2# A tibble: 10 × 3
pid group measurement
<chr> <chr> <int>
1 P1 CON 21
2 P2 CON 22
3 P3 CON 23
4 P4 CON 24
5 P5 CON 25
6 P6 TRT 26
7 P7 TRT 27
8 P8 TRT 28
9 P9 TRT 29
10 P10 TRT 30Datenframes sind wie Vektoren zentral in der Arbeit mit R und es ist deshalb wiederum wichtig sich schnell in diesen Datentyp einzuarbeiten. Tatsächlich werden ihr feststellen, dass ihr sehr selten Dataframes von Hand erstellt, sondern meistens liegen die Daten schon in digitaler Form auf eurem Rechner vor und ihr ladet mittels Funktionen, die wir später kennenlernen, die Daten in R wo sie dann als Dataframe repräsentiert werden.
Datenframes und Tibbles sind basieren tatsächlich auf einem weiteren Datentypen den Listen. Listen sind verallgemeinerte Vektoren die nicht nur einen einzigen Datentyp enthalten dürfen sondern beliebige ähnlich wie die Datentypen. Allerdings können die Elemente auch unterschiedlich lange Vektoren sein, während bei Datentypen alle Spalten die gleiche Anzahl an Elementen haben. Listen können mit der Funktion list() definiert werden.
> ls <- list(x = 1:4, y = 100, z = c('a','b'))
> ls$x
[1] 1 2 3 4
$y
[1] 100
$z
[1] "a" "b"> typeof(ls)[1] "list"Auf die einzelnen Elemente einer Liste greifen wir wiederum mit dem [[]]-Operator entweder über die Position oder bei benannten Elementen über den Bezeichner als Zeichenkette.
> ls[[1]][1] 1 2 3 4> ls[[2]][1] 100> ls[['x']][1] 1 2 3 4Wird dagegen der einfache []-Operator verwendet, dann wird das entsprechende Element zurückgegeben aber nicht als atomares Objekt sondern immer noch als Teil einer Liste.
> ls[1]$x
[1] 1 2 3 4> typeof(ls[1])[1] "list"Mit Listen lassen sich auch verschachtelte Datentypen wie z.B. Bäume konstruieren finden aber im Analysealltag relativ wenig Anwendungen. Daher werden die Listen hier auch auch nur der Vollständigkeit halber angesprochen.
Damit haben wir auch schon die wichtigsten Datentypen in R kennengelernt. In Abbildung 2.1 sind die verschiedenen Datentypen und deren Abhängigkeiten noch einmal schematisch abgebildet.
flowchart TD
A[Dataframe] --> C[Listen]
B[Matrizen] --> C
C --> D[Vektor]
D --> E(Numerisch)
D --> F(Zeichenkette)
D --> G(Logisch)
Dies war natürlich nur eine Übersicht, aber sollte euch schon relativ weit bei der Arbeit mit R bringen.