> install.packages("tidyverse")1 Eine Übersicht über R
1.1 Konzeption und Programmierparadigmen
Bei der Programmiersprache R handelt es sich um eine interpretierte Programmiersprache. Interpretiert bedeutet, dass R Programmierbefehle direkt ausführt, interpretiert. Dies steht im Gegensatz zu kompilierten Programmiersprachen (z.B. C++), bei denen gesamte Programm zunächst von einem Compiler in Maschinenbefehle übersetzt wird. Wenn eine Programmiersprache interpretiert wird, hat dies den Vorteil, dass die Arbeit besser interaktiv durchgeführt werden kann. D.h. als Anwenderin können, je nach Bedarf, einzelne Befehle oder größere zusammenhängende Befehle, an R übergeben werden und sie werden direkt ausgeführt und R liefert das Ergebnis zurück. Insbesondere bei der Datenverarbeitung vereinfacht diese Vorgehensweise die Arbeit ungemein. So können Daten per trial-and-error schnell angepasst, transformiert oder grafisch bzw. deskriptiv dargestellt werden. Dies macht den Umgang mit interpretierten Sprachen etwas intuitiver als mit kompilierten Programmiersprachen.
Der Hauptnachteil von interpretierten Programmiersprachen ist, dass durch die Abarbeitung der einzelnen Befehle bestimmte Optimierungen, um die Ausführungszeit zu verkürzen, nicht angewendet werden können. Dies hat zur Folge, dass die Ausführungszeit, d.h. die Zeit, die ein Programm benötigt, um seine Aufgaben zu erledigen, im Vergleich zu kompilierten Programmiersprachen zum Teil deutlich länger ausfallen kann. In R kann dieser Nachteil jedoch durch die Einbindung von bestimmten Paketen (was ein Paket ist, werden wir gleich sehen), die in anderen kompilierten Programmiersprachen erstellt und entsprechend optimiert wurden, in vielen Fällen umgangen werden. In vielen Fällen ist die Ausführungszeit aber tatsächlich eher zu vernachlässigen.
1.2 Ressourcen und Hilfestellungen zu R finden
War es in den Anfangszeiten von R noch teilweise schwierig, Hilfe bei auftretenden Problemen zu bekommen, hat sich dies in den letzten 10-15 Jahren glücklicherweise dramatisch geändert. Durch die etwas unglückliche Namensgebung R gab es beispielsweise zunächst Probleme bei der Suche nach Problemlösungen. Die Suchmaschinen konnten mit dem Buchstaben R wenig anfangen. Dies spielt aber heute keine Rolle mehr. Im Internet finden sich jetzt allerdings zahllose überaus aktive Communities rund um R mit Blogs, Podcasts, YouTube-Sammlungen und Programmierhilfen für alle möglichen Fragen und Probleme. Zwei sehr gute Quellen sind dabei Stack Overflow, bei denen oft schon mittels googeln sehr gute Treffer gefunden werden, oder ChatGPT bzw. Copilot. In RStudio besteht auch die Möglichkeit, Copilot direkt über GitHub zu integrieren.
Da R im wissenschaftlichen Umfeld sich sehr großer Beliebtheit erfreut, ist zudem die Menge an Büchern um und über R in den letzten Jahren geradezu explodiert. Mit eigenen Serien zur Datenanalyse mit R (Springer Use R!, CRC The R-Series) und zahllosen weiteren wissenschaftlichen Büchern mit mindestens Code-Beispielen in R bis hin zu frei verfügbaren Sammlungen hochqualitativer, wissenschaftlicher Bücher zu R (bookdown.org) lassen sich heutzutage relativ niedrigschwellig sehr gute R-Fertigkeiten aufbauen.
1.3 R Community und Pakete
Eine treibende Kraft bei der Weiterentwicklung und Weiterverbreitung von R ist die riesige Gemeinschaft von Anwenderinnen und Programmiererinnen. Dadurch, dass R im Kern eine vollständige Programmiersprache ist, kann die Funktionalität von R ständig erweitert und individuellen Bedürfnissen angepasst werden. Neue Funktionalität wird in R im Rahmen von sogenannten Paketen (alternativ Bibliotheken) gebündelt. Durch diese Pakete können neue Befehle durch neu definierte Funktionen in R zugänglich gemacht werden.
R Pakete werden über das Comprehensive R Archive Network (kurz CRAN) verteilt. CRAN ist ein internationales Netzwerk von Webservern, auf denen R Pakete gespeichert werden und das das einfache Herunterladen aus R heraus ermöglicht. Die auf CRAN gespeicherten Pakete folgen alle einer streng definierten Struktur und durchlaufen eine Qualitätskontrolle. Die Weiterentwicklung bzw. weitere Anpassung wird durch sogenannte Maintainer (Entwickler) sichergestellt. Während die Anzahl an Zusatzpaketen am Anfang von R noch relativ übersichtlich war, liegt die derzeitige Anzahl an R Paketen auf CRAN bei 19.900 (Stand 08.2023) mit Tendenz steigend. Da sich Datenanalysen über verschiedene Disziplinen und Anwendungsfälle im Grunde genommen immer wieder ähneln, besteht daher eine hohe Wahrscheinlichkeit, dass auch für ungewöhnliche Anwendungsfälle bereits bestehende Pakete und Zusatzfunktionen in R zur Verfügung stehen. Daher besteht für den Großteil von Anwenderinnen oftmals gar nicht mehr die Notwendigkeit, kompliziertere Programmieraufgaben selbst durchzuführen. Stattdessen, durch die Suche nach einem geeigneten Paket, können auftauchende Probleme schnell gelöst werden. Dies führt ebenfalls dazu, dass die Einstiegshürde für den Umgang mit R sehr niedrig ist. Da R mittlerweile zu den Top 20 der Programmiersprachen gehört, was relativ bemerkenswert für eine Spezialsprache ist, ist zudem die Investition in R-Kenntnisse heutzutage in jedem Fall positiv zu bewerten.
1.4 R, RStudio und Positron
Heutzutage wird R in den allermeisten Fällen nur mittels einer Entwicklungsumgebung, einer sogenannten IDE (Integrated Development Environment), verwendet. Die populärste IDE stellt RStudio dar, bzw. der kürzlich erschiene Nachfolger Positron. Beide IDEs stellen eine ganze Reihe von Werkzeugen zur Verfügung die den Umgang mit R deutlich vereinfachen. R tritt dann eigentlich nur noch im Hintergrund bei der Ausführung von Befehlen in Aktion. Vor allem der Editor um Skripte zu erstellen und die einfache Möglichkeit Projekte anzulegen vereinfachen die Arbeit mit R am Anfang deutlich.
Im Rahmen des vorliegenden Kurses werden wir entsprechend auch ausschließlich mit einer IDE arbeiten und Vanilla-R ignorieren, bzw. wenn R geschrieben wird, bedeutet dies eigentlich immer die IDE. Für Anwender ist es allerdings wichtig zu wissen, dass R installiert sein muss, damit die IDE auch wirklich funktionsfähig ist.
1.5 Weiterführendes
Die im weiteren Verlauf des Skripts kommenden Beispiele und Erklärungen dienen nur dazu, um einen allerersten Überblick über die Arbeit mit R zu gewinnen. D.h. die Inhalte beschränken sich auf die Konzepte, die für den Umgang mit R im späteren Verlauf dieses Statistikkurses benötigt werden. Wie schnell ersichtlich werden wird, besteht eine der Herausforderungen, den Umgang mit R zu erlernen, darin, die notwendigen Befehle und Funktionen zu kennen. Daher werden sich die ersten Schritte mit R ähnlich dem Erlernen einer neuen Sprache anfühlen. Das Schöne ist jedoch, R eignet sich hervorragend als erste Programmiersprache. In der heutigen Zeit ist das Erlernen einer Computersprache in jedem Fall eine gute Investition in die eigene Zukunft und durch mit erscheinen von LLMs ist die Anfangshürde auch noch einmal deutlich geschrumpft.
Im Internet finden sich mittlerweile zahlreiche sehr gute Quellen um den Umgang mit R über die Inhalte in in diesem Skript hinaus zu vertiefen. Für den Einstieg sind die beiden Bücher Chambers (2008) und Dalgaard (2008) empfehlenswert, von denen es auch immer günstige gebrauchte Versionen gibt. Etwas modernere Einführungen sind in Peng (2016) und Wickham, Çetinkaya-Rundel, und Grolemund (2023) zu finden, wobei es beide Bücher auch als freie Online-Versionen gibt (peng, wickham). Überhaupt eignet sich die Seite bookdown.org als gute Anlaufstelle um Anregungen zu bekommen.
1.6 Eine Beispielanalyse in R
Um das Beispiel auf dem eigenen Rechner nachzuvollziehen, müssen ein paar Pakete installiert werden. Wenn RStudio gestartet wird, dann sieht es normalerweise wie folgt aus.
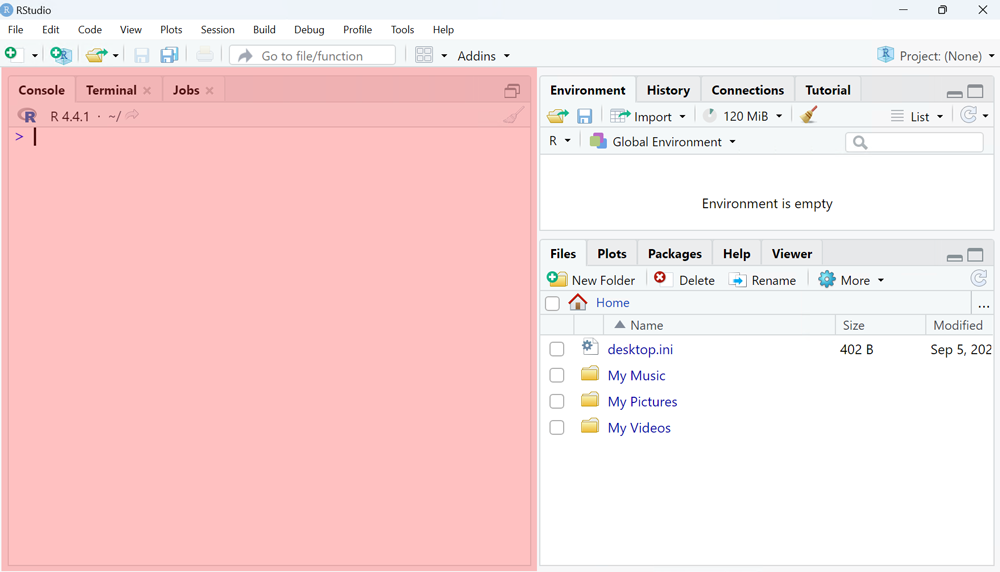
Der rote Bereich wird als Kommandozeile bezeichnet. Dort werden die Anweisungen an R übergeben. Um die benötigten Pakete zu installieren muss auf der Kommandozeile der folgende Befehl eingegeben werden.
Danach laufen eine Reihe von automatischen Befehlen durch welche die benötigten Pakete installieren.
Im Folgenden wird ein etwas Beispiel mit nur kurzen Erklärungen exemplarisch vorgeführt, um eine erste Vorstellung zu entwickeln wie die Arbeit mit R in einem konkreten Fall aussehen kann. Eingehende Erläuterungen zu den verwendeten Befehlen können in R mittels der Hilfedokumentation aufgerufen werden. Dazu wird lediglich ein ? vor den Funktionsnamen gestellt und R öffnet die zu der Funktion gehörende Hilfedatei. Soll zum Beispiel die Hilfedokumentation für die Funktion mean() aufgerufen werden. Dann muss der folgende Befehl auf der Kommandozeile eingegeben werden.
> ?meanEs sei der folgende Datensatz aus Tabelle Tabelle 1.1 gegeben. In zwei unabhängigen Gruppen A und B wurde der Körperfettgehalt bestimmt. Nun soll untersucht werden, ob ein statistisch signifikanter Unterschied zwischen den beiden Gruppen besteht. Dies ist natürlich nur ein synthetisches Beispiel und sollte in dieser Form daher nicht im Rahmen einer tatsächlichen wissenschaftlichen Arbeit durchgeführt werden, sondern dient lediglich der Anschauung wie eine solche Analyse in R durchgeführt werden könnte.
| Group | BFP |
|---|---|
| A | 13.3 |
| A | 6.0 |
| A | 20.0 |
| A | 8.0 |
| A | 14.0 |
| A | 19.0 |
| B | 22.0 |
| B | 16.0 |
| B | 21.7 |
| B | 210.0 |
| B | 30.0 |
| B | 26.0 |
| B | 30.0 |
Um eine Datenanalyse durchzuführen, müssen die Daten zunächst in R geladen werden. In der Rohform liegen die Daten in Form einer Textdatei auf dem Rechner vor. Die erste Spalte der Datei zeigt die Gruppenzugehörigkeit an, während die zweite Spalte den jeweiligen Fettgehalt beinhaltet. Die Spalten sind durch ein Komma voneinander getrennt und als Dezimaltrennzeichen, der internationalen Konvention folgend, wird ein Punkt verwendet.
Um die Daten in R zu laden wird eine spezielle Funktion aus dem Paket readr verwendet
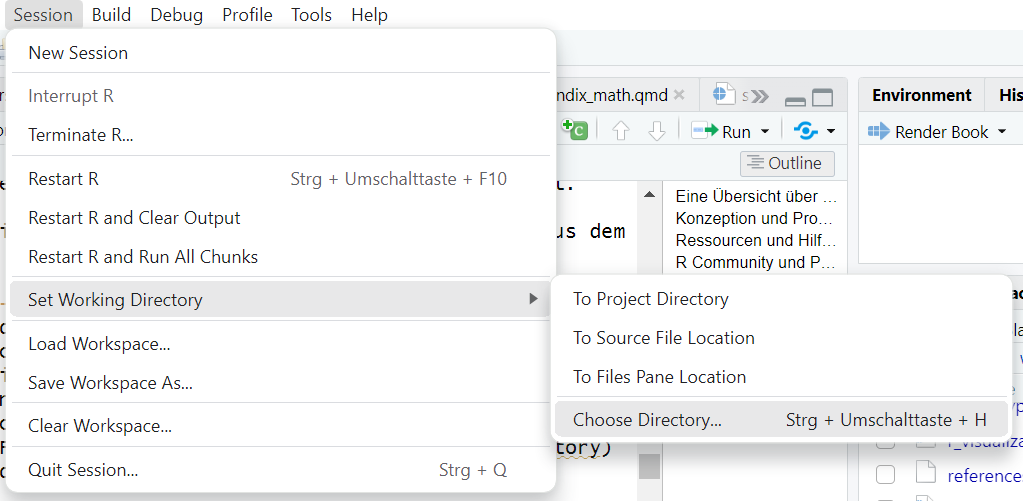
Wenn R mit dem Betriebssystem interagiert, muss immer das sogenannte Arbeitsverzeichnis (working directory) im Auge behalten werden. Das Arbeitsverzeichnis bezeichnet das Verzeichnis, das R als sein Ausgangsverzeichnis betrachtet. Wenn eine Datei von dem Computer in R eingeladen werden soll, dann muss R in der Lage sein die Datei zu finden. D.h. der Pfad muss R bekannt sein. Die Pfadangaben sind entweder in Relation zum Arbeitsverzeichnis zu geben oder absolut. Das Arbeitsverzeichnis kann mit der Funktion setwd() (kurz für set working directory) verändert werden.
In RStudio kann das Arbeitsverzeichnis über den Menüeintrag Session/Set Working Directory/Choose Directory eingestellt werden (Keyboardkürzel: SHIFT+CTRL+hSHIFT+CTRL+h).
 .
.
Die Datei sollte unten rechts im RStudio-Explorer sichtbar sein. Wenn die Datei nicht angezeigt wird, hilft meistens ein Refresh über das Zahnrad und Go To Working Directory.
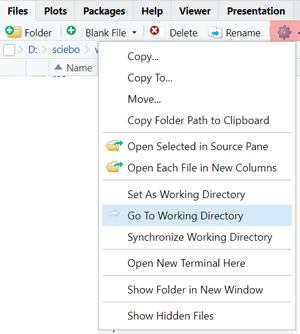
Wenn die Datei nun im Explorer in RStudio zu sehen ist, dann muss der Dateiname nicht mehr mit einem Pfad qualifiziert werden.
In Positron wird das Arbeitsverzeichnis automatisch gesetzt indem beim Start ein Ordner ausgewählt wird.
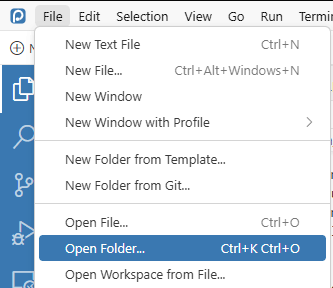
Nun wird das Paket readr geladen. readr enthält zahlreiche Funktionen zum einlesen verschiedener Dateiarten die nun in R aufgerufen werden können. Die Bespieldatei bfp_data.txt wird nun mittels der Funktion read_csv() eingelesen. read_csv() ist spezialisiert auf das Einlesen von Komma-separierten Textfiles (comma-separated-values: csv).
> library(readr)
> bfp <- read_csv(file = 'bfp_data.txt')Die Daten stehen nun unter dem Bezeichner bfp in R zur Verfügung. Der Name ist dabei wieder willkürlich gewählt und der Einfachheit halber kurz gehalten.
Die Datei mit den Beispieldaten kann unter folgendem Link heruntergeladen werden.
In R sind die geladenen Daten nun in einem sogenannten data.frame-Objekt (bzw. der neueren Version tibble) abgelegt und können nun weiterverarbeitet werden. Beispielsweise kann mittels der Funktion summary() ein Überblick über die deskriptiven Statistiken der Daten erzeugt werden.
> summary(bfp) Group BFP
Length:13 Min. : 6.00
Class :character 1st Qu.: 14.00
Mode :character Median : 20.00
Mean : 33.54
3rd Qu.: 26.00
Max. :210.00 Die Funktion summary() erzeugt für jede Datenspalte eine kurze Zusammenfassung der enthaltenen Daten. In dem Beispiel sind zwei Datenspalten vorhanden gewesen, dementsprechend erzeugt summary() zwei Ausgaben. Da in der ersten Spalte nur Buchstaben waren, wird nur eine Ausgabe über die Länge der Spalten und des “Typs” erzeugt. Dagegen wird für die zweite Spalte der Blutdruckdaten eine numerische Zusammenfassung mit Minimum, Maximum, Q1, Q3, Median und Mittelwert erzeugt.
Hier ist bereits zu sehen, dass einer der Datenpunkte wahrscheinlich fehlerhaft ist, da der Wert \(>100\) ist, was bei für einen prozentualen Körperfettanteil nicht möglich ist. Im nächsten Schritt sollen die Daten nun grafisch mittels eines Boxplots dargestellt werden. R stellt von Haus aus zahlreiche Funktionen zur einfachen graphischen Darstellung bereit. Hier soll nun das Paket ggplot2 zum Einsatz kommen, welches die Erstellung moderner Grafiken in Publikationsqualität ermöglicht (Wickham 2009). Vor der Verwendung muss das Paket ggplot2 wiederum zunächst geladen werden. Dazu wird wieder die Funktion library() verwendet.
> library(ggplot2)Nun stehen die Funktion zur Erstellung von Grafiken aus ggplot2 zur Verfügung und können entsprechend verwendet werden. Der verwendete Befehlt ist dabei erst mal nebensächlich und wird später im Verlauf des Skripts noch genauer behandelt.
> ggplot(bfp, aes(Group, BFP)) + geom_boxplot()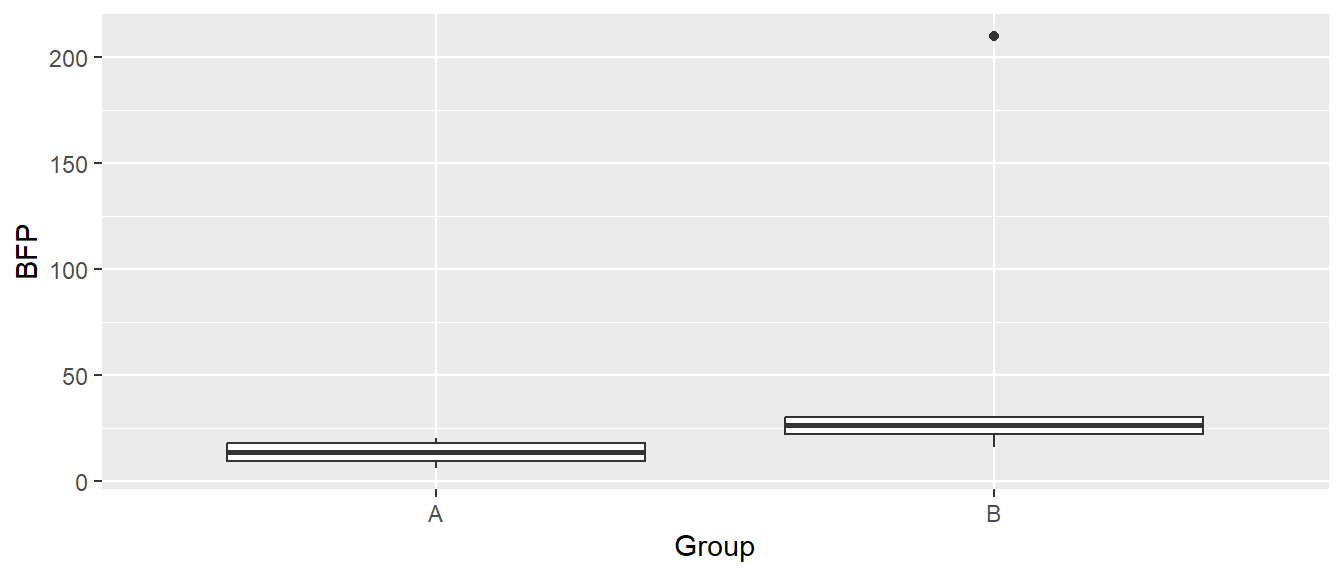
Im Boxplot in Abbildung Abbildung 1.1 ist der problematische Datenpunkt noch klarer ersichtlich und er verhindert gleichzeitig eine Analyse der Daten. Da keine weitere Information zur Verfügung stehen und klar ist, dass der Wert fehlerhaft ist, wir den Datenpunkt der Einfachheit halber ausgeschlossen. Etwas was bei tatsächlichen Daten nicht einfach so gemacht werden sollte. Um den Datenpunkte auszuschließen wird aus dem Paket dplyr die Funktion filter() verwendet. Wieder wird zunächst das Paket geladen.
> library(dplyr)Anschließend wird nun die Funktion filter() verwendet und den gefilterten Daten wird ein neuer Bezeichner bfp_clean zugewiesen.
> bfp_clean <- filter(bfp, BFP <= 100)Der Inhalt von bfp_clean kann einfach durch aufrufen des Bezeichners ausgeben werden.
> bfp_clean# A tibble: 12 × 2
Group BFP
<chr> <dbl>
1 A 13.3
2 A 6
3 A 20
4 A 8
5 A 14
6 A 19
7 B 22
8 B 16
9 B 21.7
10 B 30
11 B 26
12 B 30 Es ist zu erkennen, dass nun der fehlerhafte Wert \(>100\) fehlt. Die gesäuberten Daten können nun wieder graphisch dargestellt werden.
> ggplot(bfp_clean, aes(Group, BFP)) + geom_boxplot()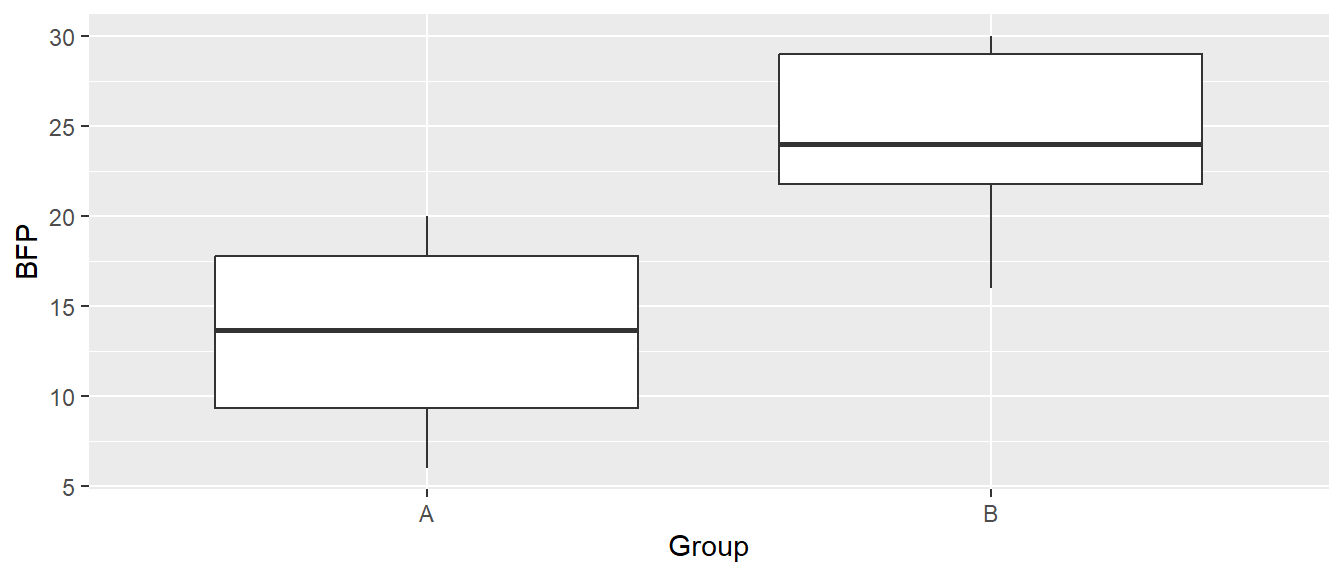
Die grafische Darstellung mittels eines Boxplots ist jetzt schon deutlich aussagekräftiger (siehe Abbildung ?fig-r-workflow-graph-02). Ohne jetzt weiter auf statistische Voraussetzungen einzugehen, kann nun ein unabhängiger t-Test für Gruppen mit unterschiedlichen Varianzen durchgeführt werden. Dazu wird wieder eine Funktion verwendet. In diesem Fall die Funktion t.test().
> t.test(BFP ~ Group, data = bfp_clean)
Welch Two Sample t-test
data: BFP by Group
t = -3.4017, df = 9.9886, p-value = 0.006762
alternative hypothesis: true difference in means between group A and group B is not equal to 0
95 percent confidence interval:
-18.040619 -3.759381
sample estimates:
mean in group A mean in group B
13.38333 24.28333 Die Ausgabe zeigt, dass ein statistisch signifikantes Ergebnis mit \(t(10) = -3.4, p = 0.007\) vorliegt.
Wie dieses Beispiel zeigt, lässt sich in R mittels weniger Befehle eine Datenanalyse realisieren. Die Entwickler von R haben dabei darauf geachtet, dass die Namensgebung von Funktionen möglichst nahe an der gewünschten Tätigkeit liegt, sodass der englische Begriff meist schnell den Namen der Funktion herleiten lässt. Im Beispiel sind alle Befehle direkt auf der Kommandozeile eingegeben worden und die Daten wurden interaktiv analysiert. Bei einer tatsächlichen Analyse wird die Datenanalyse aus einer Kombination von interaktivem Arbeiten und permanenten Skripten bestehen. Beispielsweise würden diejenigen finalen Befehle, die auf die Daten angewendet werden sollen, in eine sogenannte Skriptdatei geschrieben werden. Eine Skriptdatei ist dabei nichts anderes als eine Textdatei, in der die notwendigen Anweisungen für R untereinander stehen. Dies stellt sicher, dass die Analyse zu einem späteren Zeitpunkt wieder aufgegriffen bzw. später auch nachvollzogen werden kann. So könnte der gezeigte Beispiel-Workflow in das folgende Skript münden:
# Notwendige Bibliotheken
library(readr)
library(ggplot2)
library(dplyr)
# Daten einlesen
bfp <- read_csv(file = 'bfp_data.txt')
# Daten bearbeiten
bfp_clean <- filter(bfp, BFP <= 100)
# Deskriptiv
summary(bfp_clean)
# Graphiken
ggplot(bfp_clean, aes(Group, BFP)) + geom_boxplot()
# Analyse
t.test(BFP~Group, data = bfp_clean)Die Abfolge der Befehle in dem Skript sind durch die Verwendung von Kommentaren, die in R mit einem # signalisiert werden, noch besser nachvollziehbar. Dieses Skript könnte zusammen mit den Daten abgespeichert werden und bleibt dann ausführbar.
Das Beispiel zeigt, welche Hürden im Umgang mit R genommen werden müssen. Einmal ist es wichtig die korrekte Syntax, d.h. die Schreibregeln von R wo Klammern, Kommas und Schlüsselwörter stehen müssen, und Grammatik, die Programmstrukturregeln wie sich Anweisungen und Ausdrücke zu einem gültigen Programm zusammensetzen, zu erlernen. Dazu kommt die Kenntnis über die Funktionsnamen die im Laufe der Zeit durch wiederkehrende Benutzung sich gemerkt werden. Daher ist es wichtig so früh wie möglich, so oft wie möglich sich mit R auseinander zu setzen. Gerade am Anfang gibt es immer wieder Situationen wo es schneller geht ein Problem zum Beispiel mit Excel zu lösen. Allerdings gilt dies fast immer auch nur einmal, wenn das Problem einmal in R gelöst ist, erfolgt erfahrungsgemäß kein späterer Rückgriff aus Excel mehr.